numpy histogramの使い方を確認しながら、写真の階調性の確認のための表示に適したヒストグラムの作り方に関して考察してみようと思います。最終的にはグラフではなく画像としてヒストグラムを作ります。サンプルコードも置きました。
numpy histogram
numpy.histogramの仕様を確認し、画像のヒストグラムを取るにはどのようにしたらよいか見てみます。リファレンスより適当な和訳
numpy.histogram(a, bins=10, range=None, normed=None, weights=None, density=None)
引数
a
array_like ここで指定された配列を1次元化しヒストグラムを計算します。
bins
intの場合binの数(階級数)。
数値配列の場合にはbin境界位置。
文字列も指定可能色々指定できるようだがここでは深追いしない。
range
ビンの範囲を指定します。何も指定がないときは(a.min(), a.max())。レンジの外は無視されます。最初の値は次の値以下である必要があります。ビンの幅はこのレンジから仮にデータがなくとも、最適に(等間隔に)計算されます。
normed
bool 非推奨。正規化を目的とする場合にはdensityを使いましょう。
weight
array_like aに対して掛ける重み。aと同じサイズである必要がある。
density
bool 正規化するか否か。Falseの場合、各ビンにおけるデータ数が返されます。Trueなら、カウントの合計が1となるよう正規化された度数が返されます。bins of unity width are chosenの時は合計が1にならないそうです。なんでしょ?
返り値
hist
ヒストグラム。度数分布
bin_edges
境界位置。サイズがhistのサイズ+1になる。(先頭、終了含めた境界値が入っているため)
とのことです。
ヒストとビン
入力の配列「a」ですが、明示的に1次元と記載があります。世のサンプルコードもちゃんと引数の配列にravel()やflatten()をつけて1次元化していますが、numpy.arrayであれば、それしなくても動いているように見えます。つけた方が丁寧ですね。
やりたいことはシンプルな割に引数が分かりにくい。用語としてはbinというものを知っておく必要があります。(戻り値にもbinが出てくる。)一般的には要素数、要素そのものを意味しています。画像のヒストグラムの場合には画素の値。通常0~255の256個のbinということになります。
引数のbinには単一スカラーを渡すと全体をこの個数に等分に分割したヒストグラムを作ります。値の境界を列挙した配列(便宜上bin列と呼びます)を入れると、その範囲に収まるような度数をカウントした、ヒストグラムを作ります。等割りでないときはこちらを使うことになります。つまり、
要素0番目の度数として、bin列0番目と1番の間の値を数える。
要素1番目の度数として、bin列1番目と2番の間の値を数える。
:
要素n番目の度数として、bin列n番目とn+1番の間の値を数える。
という具合になり、bin列のサイズは、見つけたい要素数+1になる。0~255の値を集めるには、0,1,2,3…255,256までのサイズ257のbin列を用意する必要があります。
例えばこんな感じ
hist,bin = np.histogram(img,bins=np.arange(257))
257という数字がしっくりきませんがそういうものです。スカラー指定だと
hist,bin = np.histogram(img,bins=256)
でよさそうなものですが、そうもいきません。リファレンスにあるようにこの場合のデフォルトのレンジが、(a.min(), a.max())なので、この間を256割してください。という指定になっています。なので、画素値の最小値が0でない場合や、最大値が255でない場合にはbinの境界がおかしな位置になります。それを避けるためには、
hist,bin = np.histogram(img,bins=256,range=(0,255))
としてあげることになります。これで先の記述と等価になるかと思いきや、返り値のhistこそ等価ですが、bin列が異なります。先の記述だと自前で作ったbin列を引数に入れているので、整数列で0,1,2…255,256の1刻みの配列になっていますが、今回のこの記述だと、0-255までを255/256=0.99609..刻みした列が返ってきます。下図参照。
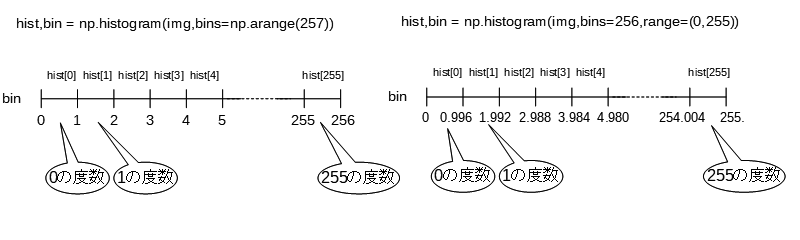
全域のヒストグラムを取りたいだけなら実害はありませんが、最後の境界値が255となっている点に違和感を感じます。基本的には、ある要素n番目の度数としてbin列n番目以上、n+1番目「未満」の数字をカウントします。そのため、最後が255では255をカウントしないのではないかと思ってしまいます。ただしリファレンスのNotesの所に、
bin列が[1,2,3,4]だったら、最初のbinは[1,2) (1以上2未満)次は[2,3)。ただし最後のbinだけは[3,4]と4を含みます。
と記載があります。最後だけ特別扱いしているようです。となると、
hist,bin = np.histogram(img,bins=np.arange(257))
この記述は256という画素値が絶対に出てこないため成立している記述で、仮に画像の中の0-127までの値の度数を取りたいときに、同じのりで、
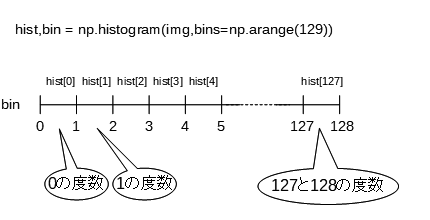
hist,bin = np.histogram(img,bins=np.arange(129))
としてしまうと、bin列は[0,1,2..127,128]となり、最後のbinには127と128の両方の画素がカウントされてしまいます。ちょっと気持ちが悪いです。
試しに、
img = np.arange(256) #0-255の画素値がそれぞれ1つずつの画像をイメージ hist,bin = np.histogram(img,bins=np.arange(129)) print(hist) [1,1,1,1,1,1,1…1,1,2]
となり、最後の要素だけ度数が2になっています。
なので、今回はrangeを指定する方式を採用したいと思います。
hist,bin = np.histogram(img,bins=256,range=(0,255))
画像のヒストグラムを取りたいだけなのに、結構引数が分かりにくいです。
正規化
引数に、normedとdensityの2つがあります。ただしnormedは非推奨とのことなので、densityの方をTrueにしてヒストグラムを取ってみます。リファレンスによると、これをTrueにすることで、得られる度数の合計が1になるようなので、つまりsum(hist)=1となるように正規化されるようです。画像で考えると画素数で割ったものが出てくる。ということになります。
ビューアーの脇に表示され、ハイライト、ダークの潰れの有無を確認する程度の用途であれば、必ずしもすべての度数を正確に知る必要はありません。むしろ正確に表示することで見にくくなっては元も子もありません。なので、用途に合わせた正規化を行う必要があります。
単純なのは、最大値で正規化する方法です。これで限られた範囲で情報の欠落なしに観察することができますが、画像によって、階調補正によってダイナミックにその形が変化してしまいます。全体の動きがつかみにくいです。また、RGBを独立に表示させる場合には、そのRGB毎に異なる最大値でそれぞれを正規化してしまうと、全体のイメージが付きにくいです。偏った、ピーキーな画像を表示させると、相対的に度数の少ない階調が分かりにくくなります。下の例だと150以上の変化や度数が分かりにくいです。
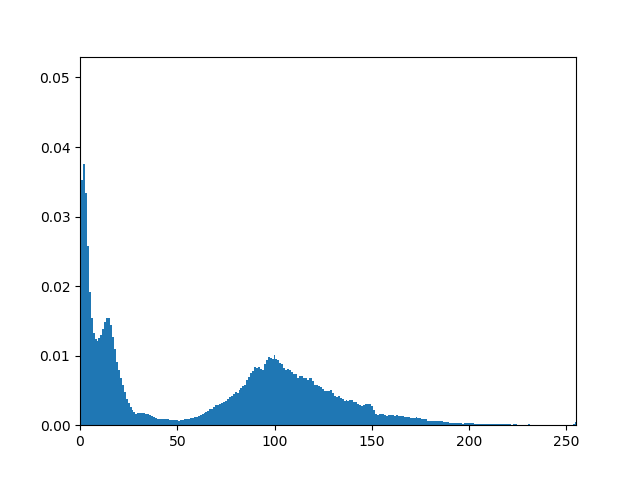
そこで、度数の最大値を画素数の1%にする。とかそんなあたりで正規化すると全体通して分かりやすくなります。度数が極端に多い階調では値がサチりますが、Photoshopのヒストグラムでもそんな感じの表示になってます。写真画像を見るにはこんなところが妥当なラインだと思います。
やり方は得られた度数のうち全体の1%、すなわち0.01より大きい値を0.01にクリップしてあげます。これで総画素数は1になりませんが、最大値0.01のヒストグラムになります。この方が写真補正時には、視覚的には見やすいです。
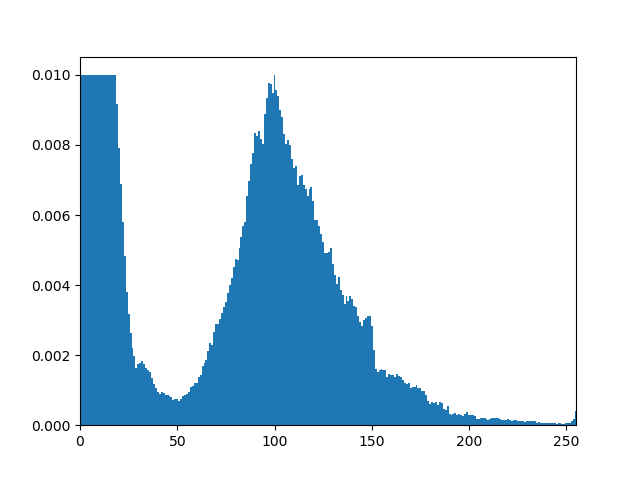
これだと、どんな画像が入力されても山のてっぺんが0.01になるので、軸もばらつかないのでその点もわかりやすいと思います。
サンプルコード
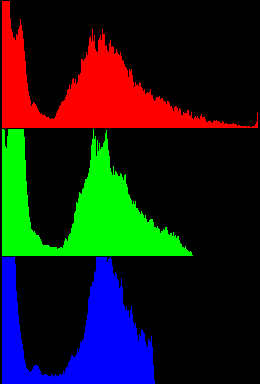
画像のヒストグラムをRGB独立に画像化するサンプルコードです。最終的にはそのRGBのヒストグラムを縦に並べた画像を作っています。
WIDTH = 260 #左右2画素は枠用 HEIGHT = 128 def createhistimg(img,color): hist, _ = np.histogram(img,bins=256, range=(0,255), density=True) hist = np.clip(hist*HEIGHT*100,0,HEIGHT-1).astype(np.uint8) #0.01をHEIGHT-1へ正規化している。 histimg = np.zeros((HEIGHT,WIDTH,3),dtype=np.uint8) for i in range(256): for j in range(hist[i]): histimg[HEIGHT-1-j][i+2] = color return histimg source = cv2.imread(FILE) hist = np.zeros((HEIGHT*3,WIDTH,3),dtype=np.uint8) hist[0 :HEIGHT, :,:] = createhistimg(source[:,:,2],[0,0,255]) hist[HEIGHT :HEIGHT*2,:,:] = createhistimg(source[:,:,1],[0,255,0]) hist[HEIGHT*2:HEIGHT*3,:,:] = createhistimg(source[:,:,0],[255,0,0])
今回は画像化させることを目的としていますが、もちろんできたヒストグラムをグラフにplotしてもよいです。こんな感じで画像化されます。
まとめ
numpy histogramの使い方をまとめました。そこからヒストグラムの画像化までを試してみました。以前書いたPython画像ビューアーのヒストグラムが貧弱だったので、それを補填するために色々調べてみました。こちらの記事のコードにもコピペした方がいいかな。




コメント